蛋白质广泛存在于生物体中的一类有机高分子,通过在特定的生理环境中形成特定的多尺度结构而发挥生理功能。综合运用分子水平计算机模拟、分子生物学、高分子物理等知识,研究蛋白质等生物大分子的结构演化过程和控制机理,已成为化学、化工、物理、生物等多学科交叉的前沿课题,对探索生命起源及医药开发具有重要意义。
近期研究方向和主要内容:
1.乳液体系的介尺度结构
基于乳液体系的实验表征,如驱油过程中阴非离子表面活性剂的耐盐性能、阴阳离子表面活性剂协同增效体系等,通过分子模拟研究不同组分间的相互作用,探索乳液体系表界面介尺度结构形成的控制机制,对其性能进行预测。并将乳液体系表界面介尺度结构的动力学模型耦合至反应器内部流体力学计算中,从而预测了油-水-表面活性剂体系在流场中的动态结构,服务于石油开采及化学工业等。
2.蛋白质分子的动态结构
生物体内的新生肽链只有折叠成具有特定三维结构(天然结构)的蛋白质分子才能获得生物活性,蛋白质如何从延展肽链折叠至此特殊结构是生物学尚待解决的重大挑战之一。分子动力学模拟作为一个强 大的模拟微观现象的方法,可以提供实验技术所不能观测到的原子尺度上及瞬时发生的微观现象,如蛋白质折叠/去折叠过程中二级结构和三级结构的转变、本质无归蛋白的动态结构、蛋白质在流场中的结构变化、蛋白质结合小分子或底物蛋白后的过程等。通过建立多尺度模型,对该过程进行机理分析,探讨蛋白质折叠机理。
3. 流感病毒的动态结构:
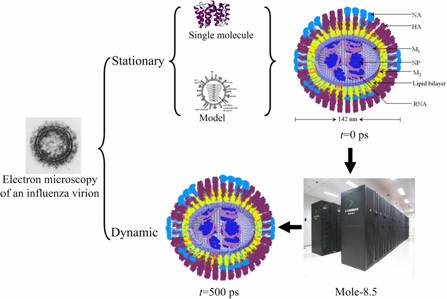
大规模分子动力学模拟流感病毒在溶液体系中的动态结构
2009年,甲型流感席卷全球,其罪魁祸首是H1N1型流感病毒。基于实验所得流感病毒的概貌,以及其主要组分(多种蛋白质、RNP等)的单分子三维结构,可通过计算机从原子水平构建流感病毒的整体结构,并采用基于GPU的动力学模拟程序计算其在生理环境下的演变。该模拟首次从原子水平复现病毒颗粒的三维动态结构,可用于流感病毒致病机理的深入分析,并进一步用于新型抗流感病毒的研制(文章链接 Ji Xu, Xiaowei Wang, Xianfeng He, Ying Ren, Wei Ge, Jinghai Li. Application of the Mole-8.5 supercomputer -- Probing the whole influenza virion at the atomic level. Chinese Science Bulletin, 2011, in press.)。
4.蛋白质复合物的研究
蛋白质同其他大分子或小分子间易形成复合物而发挥生理功能,如生物体内分子伴侣为多个蛋白质亚基形成的复杂结构,在细胞内的白质折叠过程中起关键作用,但其动态过程尚无法用实验方法进行准确测定。通过分子动力学模拟,可详细考察该过程的微观细节和控制因素。

计算机模拟底物蛋白在分子伴侣中折叠
人员队伍:任瑛,陈卫,杨瑞欣,刘佳霖
研究成果:
- Mengzhi Han, Ji Xu and Ying Ren. Sampling conformational space of intrinsically disordered proteins in explicit solvent: comparison between well-tempered ensemble approach and solute tempering method. Journal of Molecular Graphics and Modelling. 2017, 72:136-147.
- Mengzhi Han, Ji Xu and Ying Ren. Compromise in competition between free energy and binding effect of intrinsically disordered protein p53 C-Terminal Domain. Molecular Simulation, 2017, 43:110-120.
- Mengzhi Han, Ji Xu, Ying Ren, and Jinghai Li. Simulation of coupled folding and binding of an intrinsically disordered protein in explicit solvent with metadynamics. Journal of Molecular Graphics and Modelling. 2016, 68: 114-127.
- Mengzhi Han, Ji Xu, Ying Ren and Jinghai Li. Simulations of flow induced structural transition of the β-switch region of glycoprotein Ibα. Journal of Biophysical Chemistry. 2016. 209: 9–20.
- 曹天亮,韩孟之,徐骥,任瑛。驱动蛋白结构与运动机制。中国生物化学与分子生物学报。2016,32(7):734-744.
- Ji Xu, Mengzhi Han, Ying Ren and Jinghai Li. The principle of compromise in competition: exploring stability condition of protein folding. Science Bulletin. 2015 60(1):76–85
- Ji Xu, Ying Ren, Jinghai Li. Multiscale simulations of protein folding: Application to formation of secondary structures. Journal of Biomolecular Structure and Dynamics. 2013, 31(7):779-787.
- Ji Xu, Ying Ren, Wei Ge, Xiaowei Wang and Xianfeng He. A molecular view of a virus. Drug discovery and development. 2012.
http://www.dddmag.com/articles/2012/03/molecular-view-virus - Ying Ren, Jian Gao*, Ji Xu, Wei Ge and Jinghai Li. Key factors in chaperonin-assisted protein folding. Particuology. 2012, 10(1):105-116.
- Ji Xu, Xiaowei Wang, Xianfeng He, Ying Ren, Wei Ge, Jinghai Li. Application of the Mole-8.5 supercomputer -- Probing the whole influenza virion at the atomic level. Chinese Science Bulletin, 2011, 56(20):2114-2118.
- Ji Xu, Ying Ren, Wei Ge, Xiang Yu, Xizozhen Yang, Jinghai Li. Molecular Dynamics Simulation of Macromolecules Using Graphics Processing Unit. Molecular Simulation, 2010, 36(14):1131-1140.
- Ji Xu, Wei Ge, Ying Ren, Jinghai Li. Implementation of Particle-Mesh Ewald (PME) on Graphics Processing Units. Chinese Journal of Computational Physics, 2010, 27(4), 548-554.
- Jian Gao,Ying Ren and Wei Ge. Molecular dynamics simulation of effect of salt on the compromise of hydrophilic and hydrophobic interactions in sodium dodecyl sulfate micelle solutions. Chinese Journal of Chemical Engineering. 2009, 17(4):654-660.
- Ying Ren, Jian Gao, Ji Xu, Wei Ge and Jinghai Li. Explicit solvent molecular dynamics simulations of chaperonin-assisted rhodanese folding. Particuology. 2009, 7(3):220-224.
- Ying Ren, Jian Gao, Wei Ge and Jinghai Li. Thermal unfolding of rhodanese—Molecular dynamics simulation of a double-domain protein. Industrial Engineering and Chemical Research. 2009,48:8865-8871.
- Ying Ren, Jian Gao, Wei Ge, Jinghai Li and Guohua Hu. Molecular dynamics simulation of a single polymer in hydrophilic nano-slits. Chinese Science Bulletin, 2008, 53(17):2599-2606.
- Ying Ren, Wei Ge, Jian Gao and Jinghai Li. Microemulsions. In: Analytical multi-scale method for multi-phase complex systems in process engineering―Bridging reductionism and holism. Chemical Engineering Science, 2007, 62(13): 3346–3377.

